2024-11-28
导读
酶在众多生命过程中起着关键作用,准确预测酶的功能对于代谢和疾病机理研究至关重要。然而,现有的酶功能预测方法往往忽略了酶活性位点和三维结构的信息。为解决这一问题,国家超级计算深圳中心袁乾沐博士参与的科研团队研发了基于几何图学习的酶功能预测工具GraphEC,该工具利用ESMFold预测的蛋白质结构和预训练蛋白语言模型,能够高效准确地预测酶的活性位点、EC功能以及最适pH值。相关成果发表在国际顶级期刊《Nature Communications》上。该项研究由中山大学、国家超级计算深圳中心和重庆大学的多位研究人员共同完成,袁乾沐博士为该论文的共同第一作者,广州超算中心杨跃东教授为通讯作者。

引言
酶功能的准确预测一直是生物信息学领域的重点和难点,其中EC编号是定义酶功能的标准方法。然而,EC编号的实验测定过程耗时且成本高昂。因此,发展用于识别EC编号的计算方法变得尤为必要。现有的计算方法可分为基于同源序列、基于结构模板和基于机器学习的方法。前两种方法在缺少高质量的序列或结构模板时结果较差,而现有的机器学习方法依赖于人工设计的序列特征,没有考虑蛋白质的三维结构信息。为了克服这些挑战,深圳超算和中山大学的研究人员基于语言模型与几何图网络技术,开发了酶功能预测算法GraphEC。这一工具不仅提高了酶功能预测的准确性和效率,还为合成生物学、基因组学等领域的发展提供了强有力的支持。
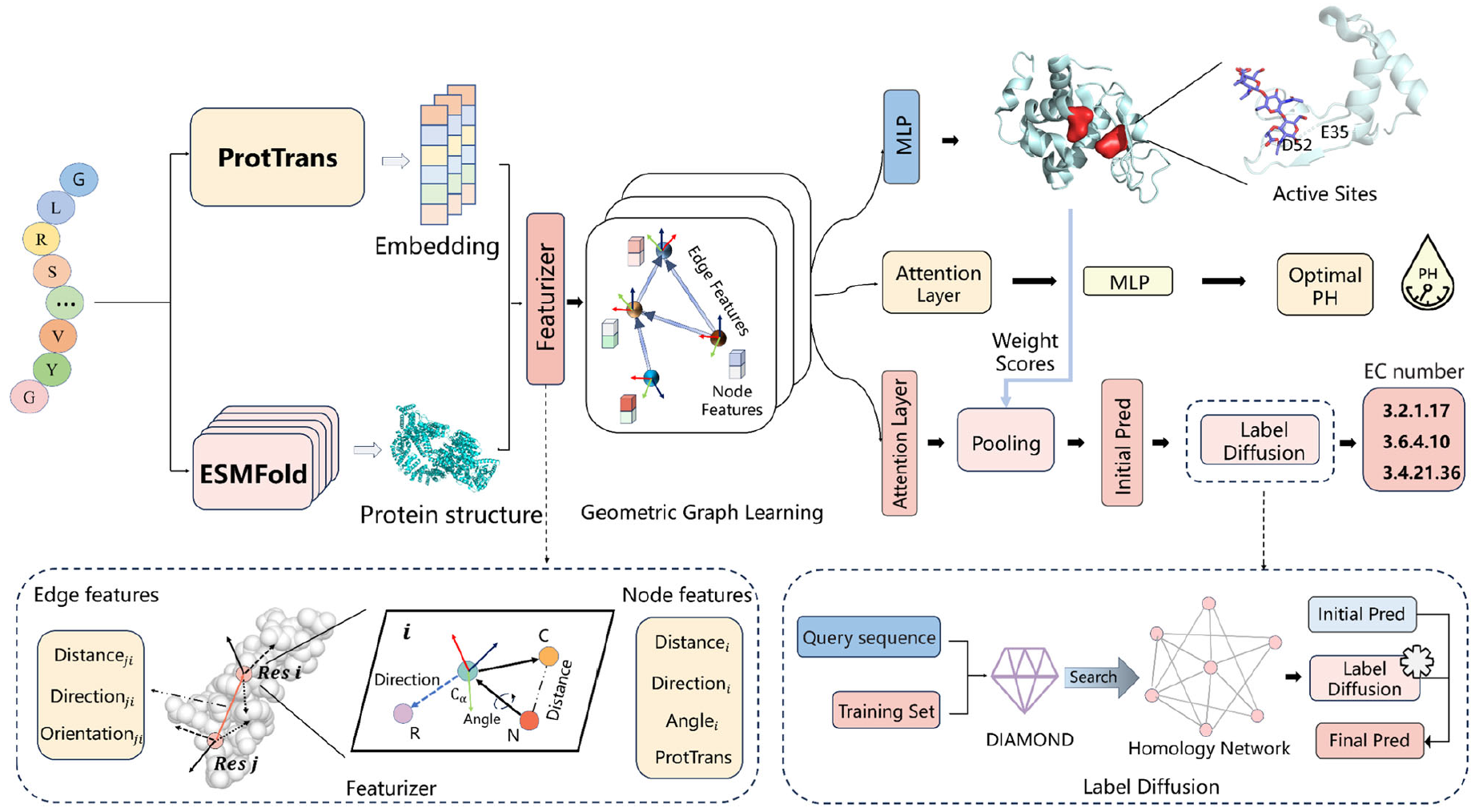
GraphEC的模型架构
GraphEC的工作流程如下:首先,给定蛋白质序列后,利用预训练的蛋白质语言模型ProtTrans提取进化信息,同时使用ESMFold预测蛋白质结构。接着,根据结构信息构建蛋白质图,并通过几何特征提取模块提取几何信息;随后,这些特征被输入到几何图学习网络中学习几何嵌入,用于预测活性位点、EC编号和反应最适pH值。具体而言,GraphEC首先预测酶的活性位点,然后利用这些信息为每个残基分配权重分数,从而指导EC编号的预测。为了提高预测的准确性,GraphEC还采用了标签扩散算法,通过提取同源信息进一步优化预测结果。最后,GraphEC还扩展到了酶反应最适pH值的预测,以进一步证明模型的通用性。
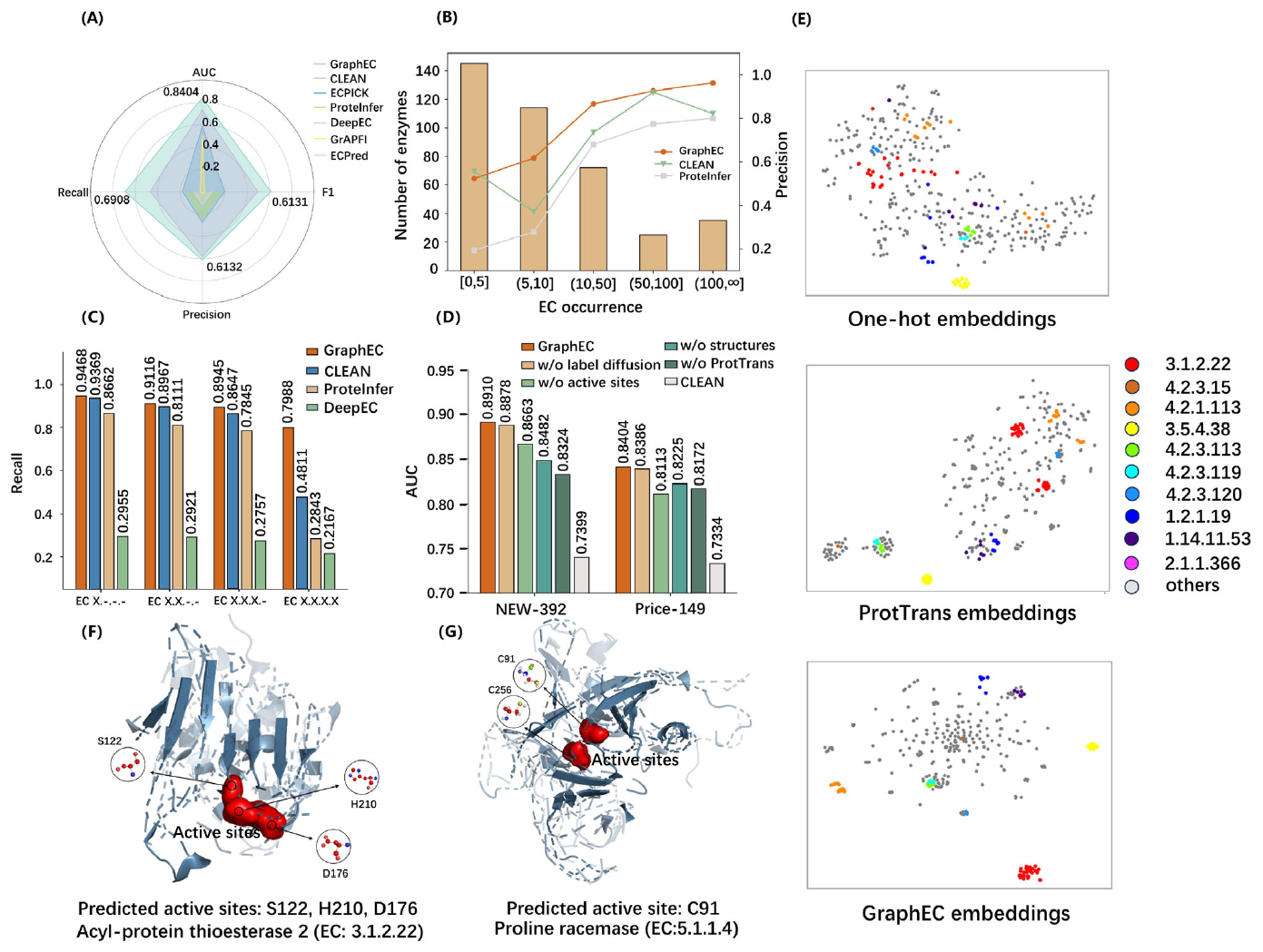
GraphEC的预测性能
研究人员在多个独立测试集上验证了GraphEC的性能。结果显示,GraphEC在预测催化活性位点、EC编号和反应最适pH值方面均优于现有最先进的方法。例如,在EC预测的Price-149数据集上,GraphEC的AUC、召回率、精确率和F1分别为0.8404、0.6908、0.6132和0.6131,比第二好的方法CLEAN(2023年发表在Science上的方法)分别提高了14.6%、47.9%、4.9%和23.9%。在NEW-392数据集上,GraphEC同样表现出色,AUC、召回率和F1得分分别为0.8910、0.7988和0.5910。同时,消融实验表明,几何结构信息与活性位点信息的引入显著地提高了模型的性能。
展望
GraphEC的成功开发标志着蛋白质功能预测领域的一个重要进展,该工具不仅能够准确预测酶的功能,还具备高效的计算能力,能够在短时间内处理大量蛋白质序列。未来,研究团队将融合超算与智算,进一步优化GraphEC,探索其在酶设计领域更多的应用场景,为新药研发、精准医疗和合成生物学等领域提供更强大的支持。
 粤公网安备44030002006735号
粤公网安备44030002006735号